 Identifiers and Description
Identifiers and Description
Gene Model Identifier
TTHERM_00648910
Standard Name
IFT52
(IntraFlagellar Transport 52 homolog)
Aliases
PreTt09205 | 94.m00123 | 3677.m00031
Description
IFT52 intraflagellar transporter-like protein; Intraflagellar transport protein; essential for ciliary assembly, maintenance, and rotokinesis; Intraflagellar transport protein 52 homolog
Genome
Browser (Macronucleus)
No Genome Browser Data Present
Genome Browser (Micronucleus)
No Genome Browser Data Present
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
Cellular Component
Biological Process
 Domains
Domains
- ( PF09822 ) ABC-type uncharacterized transport system
- ( PF21178 ) Intraflagellar transport protein 52, C-terminal domain
 Gene Expression Profile
Gene Expression Profile
 GeneMania
GeneMania
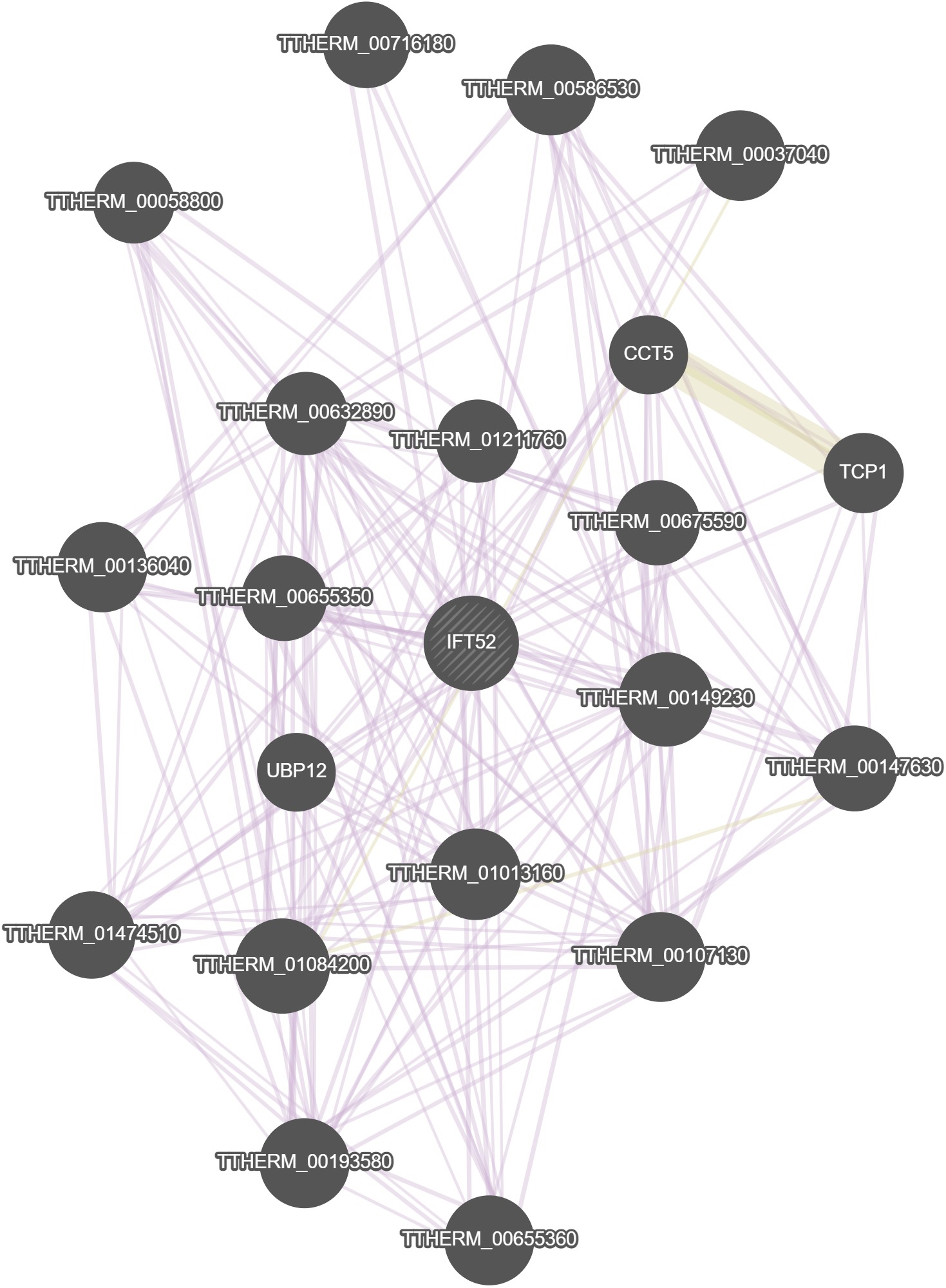
No results were calculated for this gene in GeneMania.
 Tetrahymena Stock Center
Tetrahymena Stock Center
No Data fetched for Tetrahymena Stock Center
 Homologs
Homologs
No Data fetched for Homologs
 General Information
General Information
No Data fetched for General Information
 Associated Literature
Associated Literature
- Ref:12925756: Brown JM, Fine NA, Pandiyan G, Thazhath R, Gaertig J (2003) Hypoxia regulates assembly of cilia in suppressors of Tetrahymena lacking an intraflagellar transport subunit gene. Molecular biology of the cell 14(8):3192-207
 Sequences
Sequences
>TTHERM_00648910(coding)
ATGAGTGGAGAATAGAAAATTATTGTCTTCAACGCTTCAAAGAAGGAGGCTGGTAACCCT
AGTACAAATATTAAAAAGATTATCAAGAAATATAAAGAAACATATAAGTGCGGCAGAAAT
AAAGAAGATATCACATATGATAGACTAAAGATGGCTTCTTTGGTCATATTTTTTTGCCCC
AAAGAAATGTTTACCAAGGAGGAATTCGATGCCCTCAAATAATACTTAGAAAGTGGTGGT
CGTGTCTTAGTTTTGAGCTCTGAAGGTGGTGGTCATAAGAATCGCACAAATATAAATTTC
TTCTTGGAATAATATGGTATCAGTATCAATAACGATTGTGTCGTTAGAACTGCATTTTAT
AAATACTTCCATCCTAAGGAGACTTATGTTCATTCTGGTATTTTAAATGAAGAAGTAACA
AGAGTAGCAAATGGTTTACCTAAAGAAACAAAGAGACCCCAAAATACATTCTTGTAAAAC
GTTATTGGAAAGGATGATGAAGAAGATGAATATTAAAAGGAACAATCTAGAGTTGGTTTA
GATTTTGTTTATGCCTTTGGTGCTACCTTGACTGTTTAATAACCTGCACACGCTATTTTA
GGTTCTGGTCCTCTTTCTTACCCTTCTAATAGACCAGTTTCTGCTATCGTCTAAACTAAA
AATAATGGTAGACTTGCAGTTATTGGTTCATTCGAAATGTTTACAGATGAATATTTTGAC
AATGAAGATAACTCCAAGATTTTTGATTTCTTTATAAAATATTTGCTCACAAATGAGTGC
GAATTTGAATTTAGTCCTAAAGAACCTGATGTTGAATACTTCAAGGTTCCTGATATTGCT
GAATTAGCTGATAACCTCAAGAGTTGCTTACAAGAAAGTGACCCATTACCATTTGATAGC
AAGCAATTATTTATGACAGACTTGTTTAAGTATGATGTAGACTTAGTTCCAGAAGCTGTA
AAATTGTATGAAACTCTTGGAGTAAAGCACGATCCTCTTGCTCTTATAGTTCCTTAATTC
GAAACTCCACTCCTTGGACTTGTTTCAGCTGTTTTCCCACCTATTTTAAAAGAATTAGCT
CCTCCAAGTTTAGAATTGTTTGATTTAGATGATGAATTTGCTTCAGAAAAAGTAAGACTG
GCCTAACTCACAAATAAATGCAACAACAACGATTTAGATTATTACATTAAAGAATCAGGT
GATATCTTGGGTGTAACAGATAAAGTTAAGAACAAACATGATGCCAAAGCTATTTTAAGA
TATGTTTTAGAAGAATTAATAAATTTCAAGAAGCTCAATAACTGA
>TTHERM_00648910(gene)
AAATTAATTAGTCATTGATTAATAATAGAAAAGAATCAAACAAAAAATTAAATCTTTTTA
GAAGATAGTTAGATAGATAGATACATATTACTTACTATTTCAAAGAAAAGTTAATAAATC
CGACTAGAATAAATTATAACTAACTGCAGTCATTTAGCCGATAGACATGAGTGGAGAATA
GAAAATTATTGTCTTCAACGCTTCAAAGAAGGAGGCTGGTAACCCTAGTACAAATATTAA
AAAGATTATCAAGAAATATAAAGAAACATATAAGTGCGGCAGAAATAAAGAAGATATCAC
ATATGATAGACTAAAGATGGCTTCTTTGGTCATATTTTTTTGCCCCAAAGAAATGTTTAC
CAAGGAGGAATTCGATGCCCTCAAATAATACTTAGAAAGTGGTGGTCGTGTCTTAGTTTT
GAGCTCTGAAGGTGGTGGTCATAAGAATCGCACAAATATAAATTTCTTCTTGGAATAATA
TGGTATCAGTATCAATAACGATTGTGTCGTTAGAACTGCATTTTATAAATACTTCCATCC
TAAGGAGACTTATGTTCATTCTGGTATTTTAAATGAAGAAGTAACAAGAGTAGCAAATGG
TTTACCTAAAGAAACAAAGAGACCCCAAAATACATTCTTGTAAAACGTTATTGGAAAGGA
TGATGAAGAAGATGAATATTAAAAGGAACAATCTAGAGTTGGTTTAGATTTTGTTTATGC
CTTTGGTGCTACCTTGACTGTTTAATAACCTGCACACGCTATTTTAGGTTCTGGTCCTCT
TTCTTACCCTTCTAATAGACCAGTTTCTGCTATCGTCTAAACTAAAAATAATGGTAGACT
TGCAGTTATTGGTTCATTCGAAATGTTTACAGATGAATATTTTGACAATGAAGATAACTC
CAAGATTTTTGATTTCTTTATAAAATATTTGCTCACAAATGAGTGCGAATTTGAATTTAG
TCCTAAAGAACCTGATGTTGAATACTTCAAGGTTCCTGATATTGCTGAATTAGCTGATAA
CCTCAAGAGTTGCTTACAAGAAAGTGACCCATTACCATTTGATAGCAAGCAATTATTTAT
GACAGACTTGTTTAAGTATGATGTAGACTTAGTTCCAGAAGCTGTAAAATTGTATGAAAC
TCTTGGAGTAAAGCACGATCCTCTTGCTCTTATAGTTCCTTAATTCGAAACTCCACTCCT
TGGACTTGTTTCAGCTGTTTTCCCACCTATTTTAAAAGAATTAGCTCCTCCAAGTTTAGA
ATTGTTTGATTTAGATGATGAATTTGCTTCAGAAAAAGTAAGACTGGCCTAACTCACAAA
TAAATGCAACAACAACGATTTAGATTATTACATTAAAGAATCAGGTGATATCTTGGGTGT
AACAGATAAAGTTAAGAACAAACATGATGCCAAAGCTATTTTAAGATATGTTTTAGAAGA
ATTAATAAATTTCAAGAAGCTCAATAACTGAGAATTAACTATGTATGTATTTTGTATAAA
ATAAATTAATCACTTAAAAATTAATTGCATAATATTCGAAAATATTTTTATAATTAAGCA
AGCAGAGAAAATAAACAAATCATGCTTCCATTCAACAATTAATATATTAAAACTTAACTT
GAATTAAATTTTATTGCATTCATTTCATTCTTACTTACTTATATCATCTTGTATTGGCAT
TATCAAATCTTTTTCATATTTCTTTTTTTAAATTCTTTTTGTTTAATTTAACAAAAATAA
TCTAACAACTATCAAAAATAATTTAATTTCCTATTTTTTATA
>TTHERM_00648910(protein)
MSGEQKIIVFNASKKEAGNPSTNIKKIIKKYKETYKCGRNKEDITYDRLKMASLVIFFCP
KEMFTKEEFDALKQYLESGGRVLVLSSEGGGHKNRTNINFFLEQYGISINNDCVVRTAFY
KYFHPKETYVHSGILNEEVTRVANGLPKETKRPQNTFLQNVIGKDDEEDEYQKEQSRVGL
DFVYAFGATLTVQQPAHAILGSGPLSYPSNRPVSAIVQTKNNGRLAVIGSFEMFTDEYFD
NEDNSKIFDFFIKYLLTNECEFEFSPKEPDVEYFKVPDIAELADNLKSCLQESDPLPFDS
KQLFMTDLFKYDVDLVPEAVKLYETLGVKHDPLALIVPQFETPLLGLVSAVFPPILKELA
PPSLELFDLDDEFASEKVRLAQLTNKCNNNDLDYYIKESGDILGVTDKVKNKHDAKAILR
YVLEELINFKKLNN
 Identifiers and Description
Identifiers and Description
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
 Domains
Domains
 Gene Expression Profile
Gene Expression Profile
 Vegetative Cell Cycle (Zhang et al.,
2023)
Vegetative Cell Cycle (Zhang et al.,
2023)
 GeneMania
GeneMania
 Tetrahymena Stock Center
Tetrahymena Stock Center  Homologs
Homologs
 General Information
General Information
 Associated Literature
Associated Literature
 Sequences
Sequences



