Identifiers and Description
Identifiers and Description
Gene Model Identifier
TTHERM_00118600Standard Name
PYK1 (PYruvate Kinase)Aliases
PreTt27179 | 8.m00439 | 3812.m02471Description
PYK1 pyruvate kinase complex alpha subunit; pyruvate kinase; converts phosphoenolpyruvate to pyruvate during glycolysisGenome Browser (Macronucleus)
Genome Browser (Micronucleus)
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
Molecular Function
- magnesium ion binding (IEA) | GO:0000287
- potassium ion binding (IEA) | GO:0030955
- pyruvate kinase activity (IEA) | GO:0004743
Biological Process
- glycolytic process (IEA) | GO:0006096
 Domains
Domains
No Data fetched for Domains
 Gene Expression Profile
Gene Expression Profile
 Vegetative Cell Cycle (Zhang et al.,
2023)
Vegetative Cell Cycle (Zhang et al.,
2023)
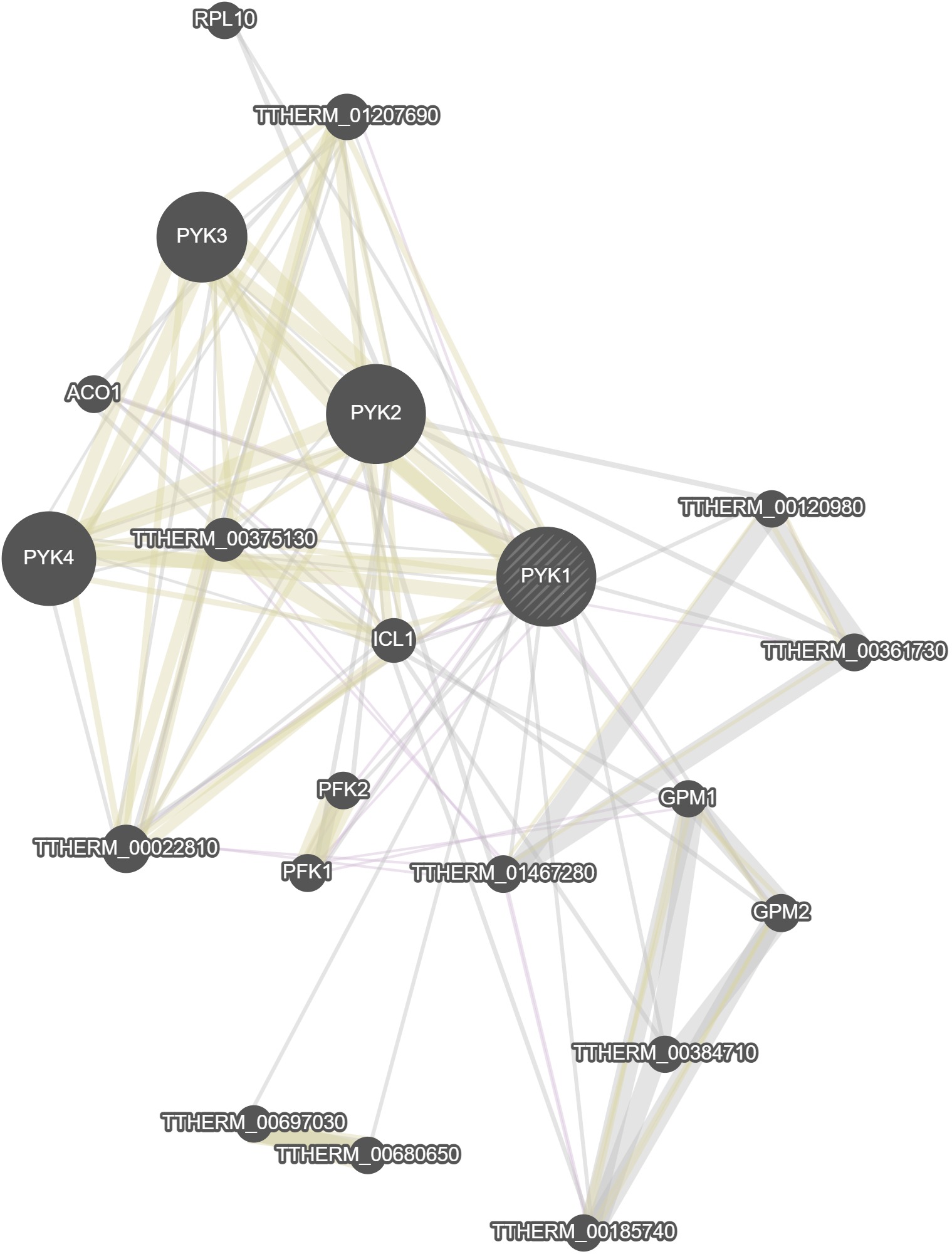
 GeneMania
GeneMania
 Tetrahymena Stock Center
Tetrahymena Stock Center
No Data fetched for Tetrahymena Stock Center
 Homologs
Homologs
Source Identifier Score Stentor Coeruleus SteCoe_6611 0 Description: None Tetrahymena borealis EI9_03875.1 9.997093137033517e-262 Description: pyruvate kinase (475 aa) Oxytricha Contig18083.0.g114 9.997093137033517e-262 Description: Pyruvate kinase, barrel domain DictyBase DDB_G0283247 4.998226778107862e-142 Description: pyk on chromosome: 4 position 344914 to 346711 WormBase WBGene00014001 8.997006431083756e-128 Description: locus:pyk-2 Pyruvate kinase status:Confirmed UniProt:Q23539 protein_id:CAA93424.2 SGD YAL038W 1.9994050701445255e-104 Description: CDC19 Pyruvate kinase, functions as a homotetramer in glycolysis to convert phosphoenolpyruvate to pyruvate, the input for aerobic (TCA cycle) or anaerobic (glucose fermentation) respiration
 General Information
General Information
No Data fetched for General Information
 Associated Literature
Associated Literature
No Data fetched for Associated Literature
 Sequences
Sequences
>TTHERM_00118600(coding)
ATGGACTCAAGCTACTCTGAATTCACTCTTGATGGTATTCTCTCCCACACTGACTACTCC
AAGAGAAAGACCAAAATTGTCTGCACTATTGGACCCTCTTGTTGGGATCACGATAATCTC
GTTTAATTGCTTGAAAATGGTATGAATGTTGCTCGTTTGAACTTCTCCCATGGTGATCAC
GCTGGTCATGGAGAAACTGTTAGAAGACTTAAGGAAGCCTTCAAAGCCAGAAAGAACATT
CAATGTGCTCTTATGCTTGATACTAAAGGACCAGAAATCAGAACTGGTTTGGTAAAAGAC
TAAACTAAGAAGCTCATCAACCTCGTTGCTGGATAAGAATTAGAAATAACTACTGACTAT
AGCGTTTTAGGAGATGAGAAAGTTCTTGCTTGCTCTTACAAAAGTTTACCCAAGAGTGTC
AAAGTTGGTGGTTAAGTCTTGATAGCCGATGGTACTTTGGTTTGCATAGTTAAGGAAATC
AAATAGGACAGTATCATCGTAAATGTTTAAAATACCTGCAGTATTGGTGAAAAGAAGAAT
ATGAATTTACCAGGTGCTATTGTTGATCTCCCTACAGTCACTGAAAAAGACGAAGATGAT
ATTGTTAACTTCGGTCTTAAGCATGGTATTGATTGTATTGCCTTGTCCTTCGCCAGAAAA
GCTGAAGATATTGAATACGTTAGAGATATCTTGGGACCTCAAGGTGAACACATTAAGATC
ATTGCAAAGATTGAAAACCAAGAAGGTCTTCATAATTATGAATAAATTTTAGATGCTGCC
GATGGTATCATGGTTGCTCGTGGTGATTTAGGTATGGAAATCCCTCCTTAGAAGGTCTTC
GTAGCTCAAAAATGGATGATCCGCAAAGCCCTTGAAAAAGGTAAACCCATTATTACTGCT
ACCTAAATGATGGAAAGTATGATTAAAAATCCCAGACCTACTAGAGCTGAAGCCAGTGAT
GTTGCTAATGCAGTTTTGGATGGTACTGATGCAGTTATGCTTTCAGGTGAAACCGCTAAC
GGATCTTTCCCTATTCAAGCTGTCTAAACCATGGCTTACATTTGCTCAGAGGCCGAACTT
TGCTATGACAACAGACAAACTTTCTGGTAAAGAACAAACAACAAGAAAAAAGTTTCTGCT
GTTGAATCTATGGCTATCTCTGCTGTCTAAATGAGTTTTGAAATTGAAAGTCCTGTTATC
ATCGTATTTACTACTAATGGTGATATGGCTAGATATGTCTCCAAGTACAGACCAAGTGCT
TAAATTTTCGTTGTATCAACTGAAAACGGTACAATTAAAGGCCTTTGCACTACTTTTGGT
GTCAGATGCTTAAGAGTCCCTTCATTCTAAGGTATTAATAAGCTCATTGATTATGCTGTT
GATGCTGCTAAAGAGTAAGGATTTATCAAGTCAGGATAAAATGCAATTGTAATCCTTGGC
TCAAACGAAGAAGAACCTGATCAAGGTGACATCCTTAAAATCAAGGAAGTTAAATGA>TTHERM_00118600(gene)
ATGGACTCAAGCTACTCTGAATTCACTCTTGATGGTATTCTCTCCCACACTGACTACTCC
AAGAGAAAGACCAAAATTGTCTGCACTATTGGGTAACCAGCAAATCAATTTTATATTATT
CATATATCTTTTGACATCCTTTCATGATTGTGTTTGCTAACAATTTGTTTTAATAATATA
GACCCTCTTGTTGGGATCACGATAATCTCGTTTAATTGCTTGAAAATGGTATGAATGTTG
CTCGTTTGAACTTCTCCCATGGTGATCACGCTGTAAGCTTCTAAAATATATTATTATTTT
GCAAAAAAGCACAATTTCAAGCTTATTAAATGACATTTTTGCTTACTATCTCCCATATTT
TAATCATAATAGGGTCATGGAGAAACTGTTAGAAGACTTAAGGAAGCCTTCAAAGCCAGA
AAGAACATTCAATGTGCTCTTATGCTTGATACTAAAGTACGTCATATTTTATACTTCACA
GCCCTTTAATAGCCCTAAAATTAATATTTTCAAAAGATTTCAATCAATTTTTTTCTAAAT
TATACTATTTATAATCTAAAAGGGACCAGAAATCAGAACTGGTTTGGTAAAAGACTAAAC
TAAGAAGCTCATCAACCTCGTTGCTGGATAAGAATTAGAAATAAGTAAAGCTTAGTATTA
AGTATTAATGAAATAAATATCATTTTTCTTTTTTAATTGTAATTTTCTAGCTACTGACTA
TAGCGTTTTAGGAGATGAGAAAGTTCTTGCTTGCTCTTACAAAAGTTTACCCAAGAGTGT
CAAAGTTGGTGGTTAAGTCTTGATAGCCGATGGTACTTTGGTTTGCATAGTTAAGGAAAT
CAAATAGGTAAAATATTAATTAGTCTTATTTAAAAAAACGTTTTTTATTTGCGAAACAAA
CAACAACAATAATAAAAATAAATAAGCATAAAGAAACTGATAGAACTTTTTAATGAAAAT
AAATAAAGAGTTTGTTTTTTATCTATAATCAGAGATAGTTAATTAATTTAAAATAAAAGC
CAAATCAATAAAAATACAATTTATTTATTGTTGGTTATTAAATTTTGATTCTGATCACAA
AAAAATTAATTATAAAAATATTTACAAAGCAAGATATTAAACTTAATTTATTGAGAATAA
TCAATCCCACTTAATAATTATTAATGCTTTAATGAATGAATGCAATTAATTTCTTCTTTT
TAACAATACAATAGGACAGTATCATCGTAAATGTTTAAAATACCTGCAGTATTGGTGAAA
AGAAGAATATGAATTTACCAGGTGCTATTGTTGATCTCCCTACAGTCACTGAAAAAGACG
AAGATGATATTGTTAACTTCGGTCTTAAGCATGGTATTGATTGTATTGCCTTGTCCTTCG
CCAGAAAAGCTGAAGATATTGAATACGTTAGAGATATCTTGGGACCTCAAGGTGAACACA
TTAAGATCATTGCAAAGATTGAAAACCAAGAAGGTCTTCATAATTATGAATAAATTTTAG
ATGCTGCCGATGGTATCATGGTTGCTCGTGGTGATTTAGGTATGGAAATCCCTCCTTAGA
AGGTCTTCGTAGCTCAAAAATGGATGATCCGCAAAGCCCTTGAAGTAATTTTAATACTAC
CTATCTATTTACTAATTTATTTTCTAATAAACAGAAAGGTAAACCCATTATTACTGCTAC
CTAAATGATGGAAAGTATGATTAAAAATCCCAGACCTACTAGAGCTGAAGCCAGTGATGT
TGCTAATGCAGTTTTGGATGGTACTGATGCAGTTATGCTTTCAGGTGAAACCGCTAACGG
ATCTTTCCCTATTCAAGCTGTCTAAACCATGGCTTACATTTGCTCAGAGGCCGAACTTTG
CTATGACAACAGACAAACTTTCTGGTAAAGAACAAACAACAAGGTATTTATTTTTATTTT
ATAGAATTAAAAATAAAATTTAGATAAGATTAACATACAAAAAGGATTTTTATCAAAATA
AAGAAAGATACAATTTGCAAATTTATTTTAGTTAGATATTATTAAACAAACTTGAATTAT
TTGTTTAGCATAAATAACATAAACATTATTTACAAAGCTAACACAAAATATTTATGATAT
ATAAAGATTCTTAAAATTAATATTTTTATGTTAAATTAATATTAAAAATAGAAAAAAGTT
TCTGCTGTTGAATCTATGGCTATCTCTGCTGTCTAAATGAGTTTTGAAATTGAAAGTCCT
GTTATCATCGTATTTACTACTAATGGTGATATGGCTAGATATGTCTCCAAGTACAGACCA
AGTGCTTAAATTTTCGTTGTATCAACTGAAAACGGTACAATTAAAGGCCTTTGCACTACT
TTTGGTGTCAGATGCTTAAGAGTCCCTTCATTCTAAGGTGAAAATTACTATTTTAACTGT
AAAATAAATATTAACTATATTCATATTAATAGGTATTAATAAGCTCATTGATTATGCTGT
TGATGCTGCTAAAGAGTAAGGATTTATCAAGTCAGGATAAAATGCAATTGTAATCCTTGG
CTCAAACGAAGAAGAACCTGATCAAGGTGACATCCTTAAAATCAAGGAAGTTAAATGA>TTHERM_00118600(protein)
MDSSYSEFTLDGILSHTDYSKRKTKIVCTIGPSCWDHDNLVQLLENGMNVARLNFSHGDH
AGHGETVRRLKEAFKARKNIQCALMLDTKGPEIRTGLVKDQTKKLINLVAGQELEITTDY
SVLGDEKVLACSYKSLPKSVKVGGQVLIADGTLVCIVKEIKQDSIIVNVQNTCSIGEKKN
MNLPGAIVDLPTVTEKDEDDIVNFGLKHGIDCIALSFARKAEDIEYVRDILGPQGEHIKI
IAKIENQEGLHNYEQILDAADGIMVARGDLGMEIPPQKVFVAQKWMIRKALEKGKPIITA
TQMMESMIKNPRPTRAEASDVANAVLDGTDAVMLSGETANGSFPIQAVQTMAYICSEAEL
CYDNRQTFWQRTNNKKKVSAVESMAISAVQMSFEIESPVIIVFTTNGDMARYVSKYRPSA
QIFVVSTENGTIKGLCTTFGVRCLRVPSFQGINKLIDYAVDAAKEQGFIKSGQNAIVILG
SNEEEPDQGDILKIKEVK