Gene Model Identifier
TTHERM_00118600
Standard Name
PYK1
(PYruvate Kinase)
Aliases
PreTt27179 | 8.m00439 | 3812.m02471
Description
PYK1 pyruvate kinase complex alpha subunit; pyruvate kinase; converts phosphoenolpyruvate to pyruvate during glycolysis; Pyruvate kinase
Genome
Browser (Macronucleus)
No Genome Browser Data Present
Genome Browser (Micronucleus)
No Genome Browser Data Present
>TTHERM_00118600(coding)
ATGGACTCAAGCTACTCTGAATTCACTCTTGATGGTATTCTCTCCCACACTGACTACTCC
AAGAGAAAGACCAAAATTGTCTGCACTATTGGACCCTCTTGTTGGGATCACGATAATCTC
GTTTAATTGCTTGAAAATGGTATGAATGTTGCTCGTTTGAACTTCTCCCATGGTGATCAC
GCTGGTCATGGAGAAACTGTTAGAAGACTTAAGGAAGCCTTCAAAGCCAGAAAGAACATT
CAATGTGCTCTTATGCTTGATACTAAAGGACCAGAAATCAGAACTGGTTTGGTAAAAGAC
TAAACTAAGAAGCTCATCAACCTCGTTGCTGGATAAGAATTAGAAATAACTACTGACTAT
AGCGTTTTAGGAGATGAGAAAGTTCTTGCTTGCTCTTACAAAAGTTTACCCAAGAGTGTC
AAAGTTGGTGGTTAAGTCTTGATAGCCGATGGTACTTTGGTTTGCATAGTTAAGGAAATC
AAATAGGACAGTATCATCGTAAATGTTTAAAATACCTGCAGTATTGGTGAAAAGAAGAAT
ATGAATTTACCAGGTGCTATTGTTGATCTCCCTACAGTCACTGAAAAAGACGAAGATGAT
ATTGTTAACTTCGGTCTTAAGCATGGTATTGATTGTATTGCCTTGTCCTTCGCCAGAAAA
GCTGAAGATATTGAATACGTTAGAGATATCTTGGGACCTCAAGGTGAACACATTAAGATC
ATTGCAAAGATTGAAAACCAAGAAGGTCTTCATAATTATGAATAAATTTTAGATGCTGCC
GATGGTATCATGGTTGCTCGTGGTGATTTAGGTATGGAAATCCCTCCTTAGAAGGTCTTC
GTAGCTCAAAAATGGATGATCCGCAAAGCCCTTGAAAAAGGTAAACCCATTATTACTGCT
ACCTAAATGATGGAAAGTATGATTAAAAATCCCAGACCTACTAGAGCTGAAGCCAGTGAT
GTTGCTAATGCAGTTTTGGATGGTACTGATGCAGTTATGCTTTCAGGTGAAACCGCTAAC
GGATCTTTCCCTATTCAAGCTGTCTAAACCATGGCTTACATTTGCTCAGAGGCCGAACTT
TGCTATGACAACAGACAAACTTTCTGGTAAAGAACAAACAACAAGAAAAAAGTTTCTGCT
GTTGAATCTATGGCTATCTCTGCTGTCTAAATGAGTTTTGAAATTGAAAGTCCTGTTATC
ATCGTATTTACTACTAATGGTGATATGGCTAGATATGTCTCCAAGTACAGACCAAGTGCT
TAAATTTTCGTTGTATCAACTGAAAACGGTACAATTAAAGGCCTTTGCACTACTTTTGGT
GTCAGATGCTTAAGAGTCCCTTCATTCTAAGGTATTAATAAGCTCATTGATTATGCTGTT
GATGCTGCTAAAGAGTAAGGATTTATCAAGTCAGGATAAAATGCAATTGTAATCCTTGGC
TCAAACGAAGAAGAACCTGATCAAGGTGACATCCTTAAAATCAAGGAAGTTAAATGA
>TTHERM_00118600(gene)
CTTTAATTAAAATCTCTTAAAATAAATAAATAAACAATATATAAATGGACTCAAGCTACT
CTGAATTCACTCTTGATGGTATTCTCTCCCACACTGACTACTCCAAGAGAAAGACCAAAA
TTGTCTGCACTATTGGACCCTCTTGTTGGGATCACGATAATCTCGTTTAATTGCTTGAAA
ATGGTATGAATGTTGCTCGTTTGAACTTCTCCCATGGTGATCACGCTGGTCATGGAGAAA
CTGTTAGAAGACTTAAGGAAGCCTTCAAAGCCAGAAAGAACATTCAATGTGCTCTTATGC
TTGATACTAAAGGACCAGAAATCAGAACTGGTTTGGTAAAAGACTAAACTAAGAAGCTCA
TCAACCTCGTTGCTGGATAAGAATTAGAAATAACTACTGACTATAGCGTTTTAGGAGATG
AGAAAGTTCTTGCTTGCTCTTACAAAAGTTTACCCAAGAGTGTCAAAGTTGGTGGTTAAG
TCTTGATAGCCGATGGTACTTTGGTTTGCATAGTTAAGGAAATCAAATAGGACAGTATCA
TCGTAAATGTTTAAAATACCTGCAGTATTGGTGAAAAGAAGAATATGAATTTACCAGGTG
CTATTGTTGATCTCCCTACAGTCACTGAAAAAGACGAAGATGATATTGTTAACTTCGGTC
TTAAGCATGGTATTGATTGTATTGCCTTGTCCTTCGCCAGAAAAGCTGAAGATATTGAAT
ACGTTAGAGATATCTTGGGACCTCAAGGTGAACACATTAAGATCATTGCAAAGATTGAAA
ACCAAGAAGGTCTTCATAATTATGAATAAATTTTAGATGCTGCCGATGGTATCATGGTTG
CTCGTGGTGATTTAGGTATGGAAATCCCTCCTTAGAAGGTCTTCGTAGCTCAAAAATGGA
TGATCCGCAAAGCCCTTGAAAAAGGTAAACCCATTATTACTGCTACCTAAATGATGGAAA
GTATGATTAAAAATCCCAGACCTACTAGAGCTGAAGCCAGTGATGTTGCTAATGCAGTTT
TGGATGGTACTGATGCAGTTATGCTTTCAGGTGAAACCGCTAACGGATCTTTCCCTATTC
AAGCTGTCTAAACCATGGCTTACATTTGCTCAGAGGCCGAACTTTGCTATGACAACAGAC
AAACTTTCTGGTAAAGAACAAACAACAAGAAAAAAGTTTCTGCTGTTGAATCTATGGCTA
TCTCTGCTGTCTAAATGAGTTTTGAAATTGAAAGTCCTGTTATCATCGTATTTACTACTA
ATGGTGATATGGCTAGATATGTCTCCAAGTACAGACCAAGTGCTTAAATTTTCGTTGTAT
CAACTGAAAACGGTACAATTAAAGGCCTTTGCACTACTTTTGGTGTCAGATGCTTAAGAG
TCCCTTCATTCTAAGGTATTAATAAGCTCATTGATTATGCTGTTGATGCTGCTAAAGAGT
AAGGATTTATCAAGTCAGGATAAAATGCAATTGTAATCCTTGGCTCAAACGAAGAAGAAC
CTGATCAAGGTGACATCCTTAAAATCAAGGAAGTTAAATGATCTAAAAAAAATATAATAC
CAATTTATAATATTTTACTTTAATTTATTTGAAAATTATGTAATCACTCTTCATAGTATT
GTAAAGTGTTAAGCTTGTTAAGCAATAAAATCTTTAAAACT
>TTHERM_00118600(protein)
MDSSYSEFTLDGILSHTDYSKRKTKIVCTIGPSCWDHDNLVQLLENGMNVARLNFSHGDH
AGHGETVRRLKEAFKARKNIQCALMLDTKGPEIRTGLVKDQTKKLINLVAGQELEITTDY
SVLGDEKVLACSYKSLPKSVKVGGQVLIADGTLVCIVKEIKQDSIIVNVQNTCSIGEKKN
MNLPGAIVDLPTVTEKDEDDIVNFGLKHGIDCIALSFARKAEDIEYVRDILGPQGEHIKI
IAKIENQEGLHNYEQILDAADGIMVARGDLGMEIPPQKVFVAQKWMIRKALEKGKPIITA
TQMMESMIKNPRPTRAEASDVANAVLDGTDAVMLSGETANGSFPIQAVQTMAYICSEAEL
CYDNRQTFWQRTNNKKKVSAVESMAISAVQMSFEIESPVIIVFTTNGDMARYVSKYRPSA
QIFVVSTENGTIKGLCTTFGVRCLRVPSFQGINKLIDYAVDAAKEQGFIKSGQNAIVILG
SNEEEPDQGDILKIKEVK
 Identifiers and Description
Identifiers and Description
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
 Domains
Domains
 Gene Expression Profile
Gene Expression Profile
 Vegetative Cell Cycle (Zhang et al.,
2023)
Vegetative Cell Cycle (Zhang et al.,
2023)
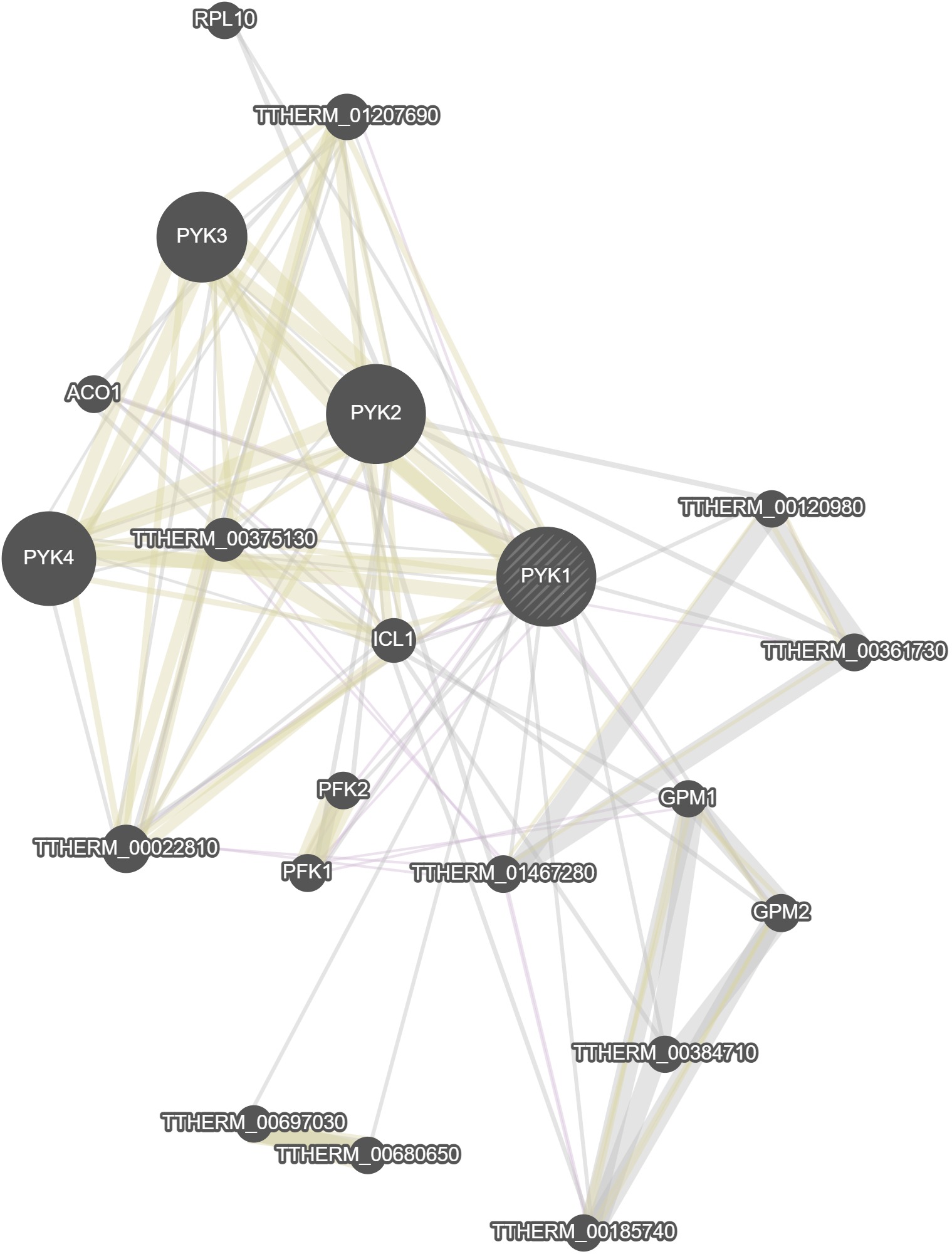
 GeneMania
GeneMania
 Tetrahymena Stock Center
Tetrahymena Stock Center  Homologs
Homologs
 General Information
General Information
 Associated Literature
Associated Literature
 Sequences
Sequences