Gene Model Identifier
TTHERM_00218330
Standard Name
VMA16
(Vacuolar Membrane Atpase)
Aliases
PreTt22260 | 18.m00286 | 3701.m00046
Description
VMA16 vacuolar proton-ATPase subunit C proteolipid protein; ATP synthase subunit C family protein; V-ATPase proteolipid subunit C-like domain
Genome
Browser (Macronucleus)
No Genome Browser Data Present
Genome Browser (Micronucleus)
No Genome Browser Data Present
>TTHERM_00218330(coding)
ATGGCTGATTACTAATTTTAAGATAATTGGAACGATTTATTCGCTTCCATTAGTGGTTAT
TCATGGGCTTACTTAGGTATTGGTTTAGCTCTTGGTACCTCCATTGCAGGTGCTGCCTGG
GGTATTTTCATTACTGGTGCTTCTCTTTTGGGTGCTTCAGTCAAGGCTCCAAGTATTAGA
TCTAAGAATTTGATCAGTATCATTTTCTGTGAAGCCGTTGCTATTTATGGTGTAATTATG
GCCATTATTATGTAAGGTAAAATGGCCAATTTTACAGGTGGTATTCCTAATGATAATTAA
ATATTCTTGATCCAATATTCAGGCTATGCTATTTTCTGGACTGGTATCTCTGTAGGTATC
TCTAACTTTGTTTGTGGTATTTGTGTCGGTATTACTGGTAGTGGTTGTGCTATTTCTCAT
GCTCAATAAGCTGAAACCTTCGTTAAAATTTTGGTTATTGAAATTTTTGGAAGCGCTCTC
GGTCTTTTCGGTGTTATTGTAGGTATTATCTAATGTGGTGATGTTTCTTTCCCTACAAAG
TGA
>TTHERM_00218330(gene)
ATATCATCAATCAATTTAATCTAAAAAGAAAAAGAAAAAAAAATAATATCATGGCTGATT
ACTAATTTTAAGATAATTGGAACGATTTATTCGCTTCCATTAGTGGTTATTCATGGGCTT
ACTTAGGTATTGGTTTAGCTCTTGGTACCTCCATTGCAGGTGCTGCCTGGGGTATTTTCA
TTACTGGTGCTTCTCTTTTGGGTGCTTCAGTCAAGGCTCCAAGTATTAGATCTAAGAATT
TGATCAGTATCATTTTCTGTGAAGCCGTTGCTATTTATGGTGTAATTATGGCCATTATTA
TGTAAGGTAAAATGGCCAATTTTACAGGTGGTATTCCTAATGATAATTAAATATTCTTGA
TCCAATATTCAGGCTATGCTATTTTCTGGACTGGTATCTCTGTAGGTATCTCTAACTTTG
TTTGTGGTATTTGTGTCGGTATTACTGGTAGTGGTTGTGCTATTTCTCATGCTCAATAAG
CTGAAACCTTCGTTAAAATTTTGGTTATTGAAATTTTTGGAAGCGCTCTCGGTCTTTTCG
GTGTTATTGTAGGTATTATCTAATGTGGTGATGTTTCTTTCCCTACAAAGTGATCATTTA
TCATGCTAAATTGGATTATTGATTGATTTCATGAAAATAAACTAAATAATTCTAATAAAT
TTTAATAATAAAGAACTTAATCTATAAATAGTTATTTTATTGGTAGAGAAATACTTTTAA
AATGTATGGTCTATTTTCTAAAAATGTATTACTTTTTTTATGGCTAATTATAATATCGAT
TACTCATAACAATTTATACATATTTATATATACATATATCTTTCTATATATTTTCACAAA
TAAACT
>TTHERM_00218330(protein)
MADYQFQDNWNDLFASISGYSWAYLGIGLALGTSIAGAAWGIFITGASLLGASVKAPSIR
SKNLISIIFCEAVAIYGVIMAIIMQGKMANFTGGIPNDNQIFLIQYSGYAIFWTGISVGI
SNFVCGICVGITGSGCAISHAQQAETFVKILVIEIFGSALGLFGVIVGIIQCGDVSFPTK
 Identifiers and Description
Identifiers and Description
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
 Domains
Domains
 Gene Expression Profile
Gene Expression Profile
 Vegetative Cell Cycle (Zhang et al.,
2023)
Vegetative Cell Cycle (Zhang et al.,
2023)
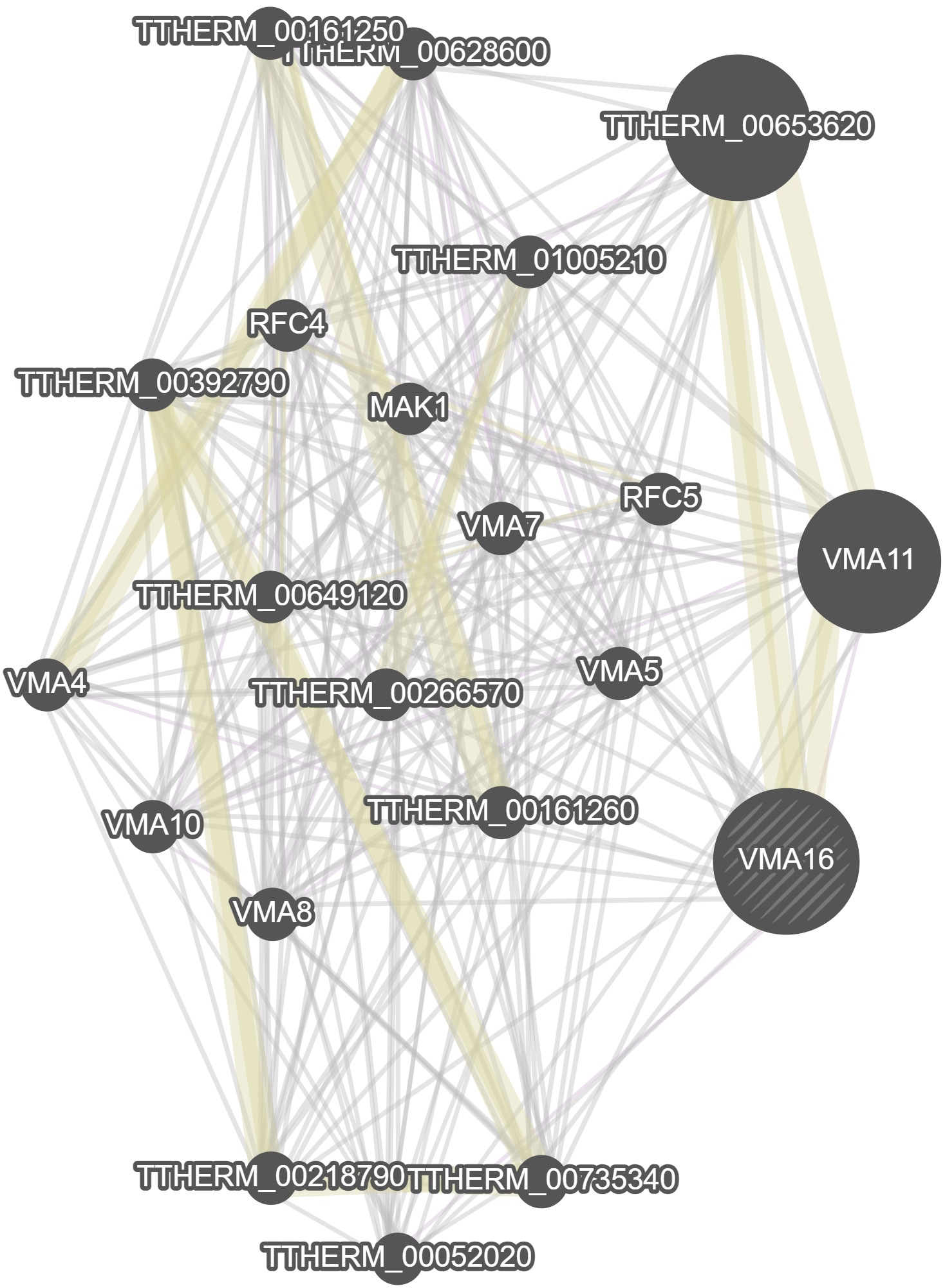
 GeneMania
GeneMania
 Tetrahymena Stock Center
Tetrahymena Stock Center  Homologs
Homologs
 General Information
General Information
 Associated Literature
Associated Literature
 Sequences
Sequences