Gene Model Identifier
TTHERM_00529860
Standard Name
VMA2
(Vacuolar Membrane Atpase)
Aliases
PreTt04655 | 67.m00170 | 3684.m00040
Description
VMA2 vacuolar V-type H(+)-ATPase B subunit; V-type ATPase, B subunit family protein; V-type ATP synthase regulatory subunit B/beta
Genome
Browser (Macronucleus)
No Genome Browser Data Present
Genome Browser (Micronucleus)
No Genome Browser Data Present
>TTHERM_00529860(coding)
ATGTCTTCTGATCCTATAGCTGCTAAGATTCACGCAGCCGCAGCATCTAGAGATTATAAT
GTTGCTCCTCGTGCTGATTACAGAACAGTTTGCAAAGTCGATGGTCCTTTGGTTATCCTC
GATAATGTCAAATTCCCCAAGTTTGCTGAAATCGTTAACGTTTGCTTAGCCGATGGTACA
ATCCGTAAAGGTTAGGTTCTCGAAATCAGTGGTAAAAAGGCCGTCGTTTAGATCTTTGAA
GGTACTGATGGTATCGATAGTTTATATACTCACTGTGAATTCACTGGTGAAACTTTATAA
ATGCCCATTGCCGAAGAAATGTTAGGTAGAGTCTTCAATGGTTCTGGTACTCCTATTGAC
AAGGGTCCACCTGTTTTGGCTGAAAAATTCTAAAGTATTAATGGTCAACCCATTAATCCT
TACTCCAGAGTTTATCCCAAGGAAATGATTCAAACTGGTATTTCTGCTATCGATTGTATG
ACTTCTATTGCTAGAGGTTAAAAGATTCCTCTTTTCTCTGCTAACGGTCTTCCCCATAAT
GAAATTGGTGCTTAAATCGTGCGTTAAGCTTCTTTGGTTAAGGGTAAGGATGTTTTAGAT
CACTCCGAAGAAAACTTCGCCGTCGTTTTCGGTGCTATGGGTGTTAATATGGAAGTCGCT
CGTTTCTTCAGATAAGATTTCGAATCTAACGGTTCCATGGAAAAGGTTATTCTTTTCCTT
AATCTTGCTAATGATCCCACTATCGAACGTATCATTACTCCTCGTCTTTCTTTGACCGCT
GCCGAATATTTAGCTTACGAAAGAGAAATGCACGTCTTGGTTATCTTAACTGATATGTCA
TAATATGCTGATTCTTTGAGAGAAGTTTCAGCTGCCAGAGAAGAAGTCCCTGGTAGAAGA
TCTTATCCTGGTTATTTATATACTGATTTATCAACAATCTACGAAAGAGCCGGTAGAGTC
GAAGGTGTTAATGGTTCCATCACTTAAATTCCCATTCTTACCATGCCTTCTGATGATATT
ACCCATCCTATTCCCGATTTGACTGGTTATATTACTGAAGGTTAAATCTTCATTGATCGT
TAATTACATAATAAGTAAATTTATCCTCCCATCAATGTCTTACCTTCTCTTTCTCGTCTC
ATGAAGTCTGCTATTGGTATTGATGATCACGGTAAGGTCATGACCAGAGTTGATCACCCT
GATGTCTCCAACGAACTCTATGCTGCTTATGCTATTGGTAAGGATACTGCTGCAATGAAG
GCTGTCGTCGGTGAAGAAGCTCTTAATGAAGAAGATCACCTTTATCTTGAATTCTTAAAG
AATTTCGAATCTAAATATATTTCTCAAGGTCAATACGAATCTAGAACTATCTTCTAAACC
TTAAACAAAGCTTGGGAATTACTCAGAATTTTCCCTCCAGAGAAGCTAAAAAAGATTAAA
CAATCAACCAAGGATTTGTACTACGCTAAAAGGGGTGAAGAAGAAGATAACTGA
>TTHERM_00529860(gene)
TATTTTTTTACTTTACTATACCTTTGTTTGGCTCATTAGTTAAGGTGGAACAAGCAATTA
TTTGGCTATAGGGATGAAATCCTAATAAAAATTTTTATTTATTATAATAAAATATAAATA
AAAAAACAAAAATAAAAATAAAAAAGAAAAAGAAGAAAAATGTCTTCTGATCCTATAGCT
GCTAAGATTCACGCAGCCGCAGCATCTAGAGATTATAATGTTGCTCCTCGTGCTGATTAC
AGAACAGTTTGCAAAGTCGATGGTCCTTTGGTTATCCTCGATAATGTCAAATTCCCCAAG
TTTGCTGAAATCGTTAACGTTTGCTTAGCCGATGGTACAATCCGTAAAGGTTAGGTTCTC
GAAATCAGTGGTAAAAAGGCCGTCGTTTAGATCTTTGAAGGTACTGATGGTATCGATAGT
TTATATACTCACTGTGAATTCACTGGTGAAACTTTATAAATGCCCATTGCCGAAGAAATG
TTAGGTAGAGTCTTCAATGGTTCTGGTACTCCTATTGACAAGGGTCCACCTGTTTTGGCT
GAAAAATTCTAAAGTATTAATGGTCAACCCATTAATCCTTACTCCAGAGTTTATCCCAAG
GAAATGATTCAAACTGGTATTTCTGCTATCGATTGTATGACTTCTATTGCTAGAGGTTAA
AAGATTCCTCTTTTCTCTGCTAACGGTCTTCCCCATAATGAAATTGGTGCTTAAATCGTG
CGTTAAGCTTCTTTGGTTAAGGGTAAGGATGTTTTAGATCACTCCGAAGAAAACTTCGCC
GTCGTTTTCGGTGCTATGGGTGTTAATATGGAAGTCGCTCGTTTCTTCAGATAAGATTTC
GAATCTAACGGTTCCATGGAAAAGGTTATTCTTTTCCTTAATCTTGCTAATGATCCCACT
ATCGAACGTATCATTACTCCTCGTCTTTCTTTGACCGCTGCCGAATATTTAGCTTACGAA
AGAGAAATGCACGTCTTGGTTATCTTAACTGATATGTCATAATATGCTGATTCTTTGAGA
GAAGTTTCAGCTGCCAGAGAAGAAGTCCCTGGTAGAAGATCTTATCCTGGTTATTTATAT
ACTGATTTATCAACAATCTACGAAAGAGCCGGTAGAGTCGAAGGTGTTAATGGTTCCATC
ACTTAAATTCCCATTCTTACCATGCCTTCTGATGATATTACCCATCCTATTCCCGATTTG
ACTGGTTATATTACTGAAGGTTAAATCTTCATTGATCGTTAATTACATAATAAGTAAATT
TATCCTCCCATCAATGTCTTACCTTCTCTTTCTCGTCTCATGAAGTCTGCTATTGGTATT
GATGATCACGGTAAGGTCATGACCAGAGTTGATCACCCTGATGTCTCCAACGAACTCTAT
GCTGCTTATGCTATTGGTAAGGATACTGCTGCAATGAAGGCTGTCGTCGGTGAAGAAGCT
CTTAATGAAGAAGATCACCTTTATCTTGAATTCTTAAAGAATTTCGAATCTAAATATATT
TCTCAAGGTCAATACGAATCTAGAACTATCTTCTAAACCTTAAACAAAGCTTGGGAATTA
CTCAGAATTTTCCCTCCAGAGAAGCTAAAAAAGATTAAACAATCAACCAAGGATTTGTAC
TACGCTAAAAGGGGTGAAGAAGAAGATAACTGATTACTAATTTTATATAAGTTTTTTAAC
GTTTATCATATCAGAGTATTAAATATAAATTTATTATACAGTAATTTTTAGTAAGCAAAG
CAAGTAAGCAAGTATCTATCTATATCATAGTATTATTCATCCTTTCAAATTCAACAAATT
AAAAAGTTCATTCACTTGTTTATTTAGTTCATATTT
>TTHERM_00529860(protein)
MSSDPIAAKIHAAAASRDYNVAPRADYRTVCKVDGPLVILDNVKFPKFAEIVNVCLADGT
IRKGQVLEISGKKAVVQIFEGTDGIDSLYTHCEFTGETLQMPIAEEMLGRVFNGSGTPID
KGPPVLAEKFQSINGQPINPYSRVYPKEMIQTGISAIDCMTSIARGQKIPLFSANGLPHN
EIGAQIVRQASLVKGKDVLDHSEENFAVVFGAMGVNMEVARFFRQDFESNGSMEKVILFL
NLANDPTIERIITPRLSLTAAEYLAYEREMHVLVILTDMSQYADSLREVSAAREEVPGRR
SYPGYLYTDLSTIYERAGRVEGVNGSITQIPILTMPSDDITHPIPDLTGYITEGQIFIDR
QLHNKQIYPPINVLPSLSRLMKSAIGIDDHGKVMTRVDHPDVSNELYAAYAIGKDTAAMK
AVVGEEALNEEDHLYLEFLKNFESKYISQGQYESRTIFQTLNKAWELLRIFPPEKLKKIK
QSTKDLYYAKRGEEEDN
 Identifiers and Description
Identifiers and Description
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
 Domains
Domains
 Gene Expression Profile
Gene Expression Profile
 Vegetative Cell Cycle (Zhang et al.,
2023)
Vegetative Cell Cycle (Zhang et al.,
2023)
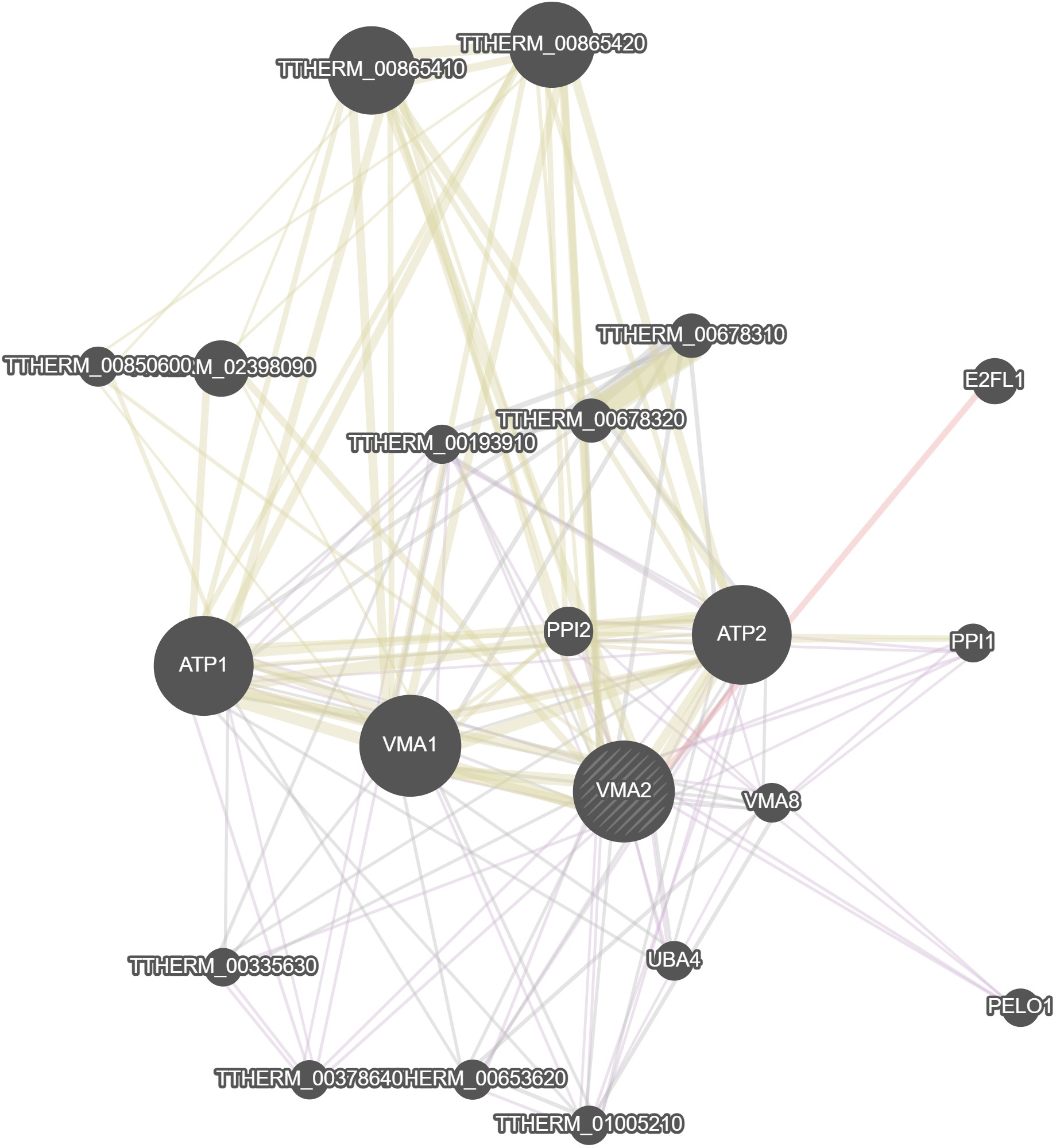
 GeneMania
GeneMania
 Tetrahymena Stock Center
Tetrahymena Stock Center  Homologs
Homologs
 General Information
General Information
 Associated Literature
Associated Literature
 Sequences
Sequences