 Identifiers and Description
Identifiers and Description
Gene Model Identifier
TTHERM_00823550
Standard Name
PRKA1
(homolog to human PRKAA1)
Aliases
PreTt20428 | 141.m00085 | 3682.m00019
Description
PRKA1 kinase domain protein; Sequence similarity to the AMPK subfamily of the protein kinase CAMKL family (Eisen et al. 2006); homolog to the catalytic subunit of the 5'-prime-AMP-activated protein kinase (AMPK); Protein kinase domain
Genome
Browser (Macronucleus)
No Genome Browser Data Present
Genome Browser (Micronucleus)
No Genome Browser Data Present
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
No Data fetched for Gene Ontology Annotations
 Domains
Domains
 Gene Expression Profile
Gene Expression Profile
 GeneMania
GeneMania
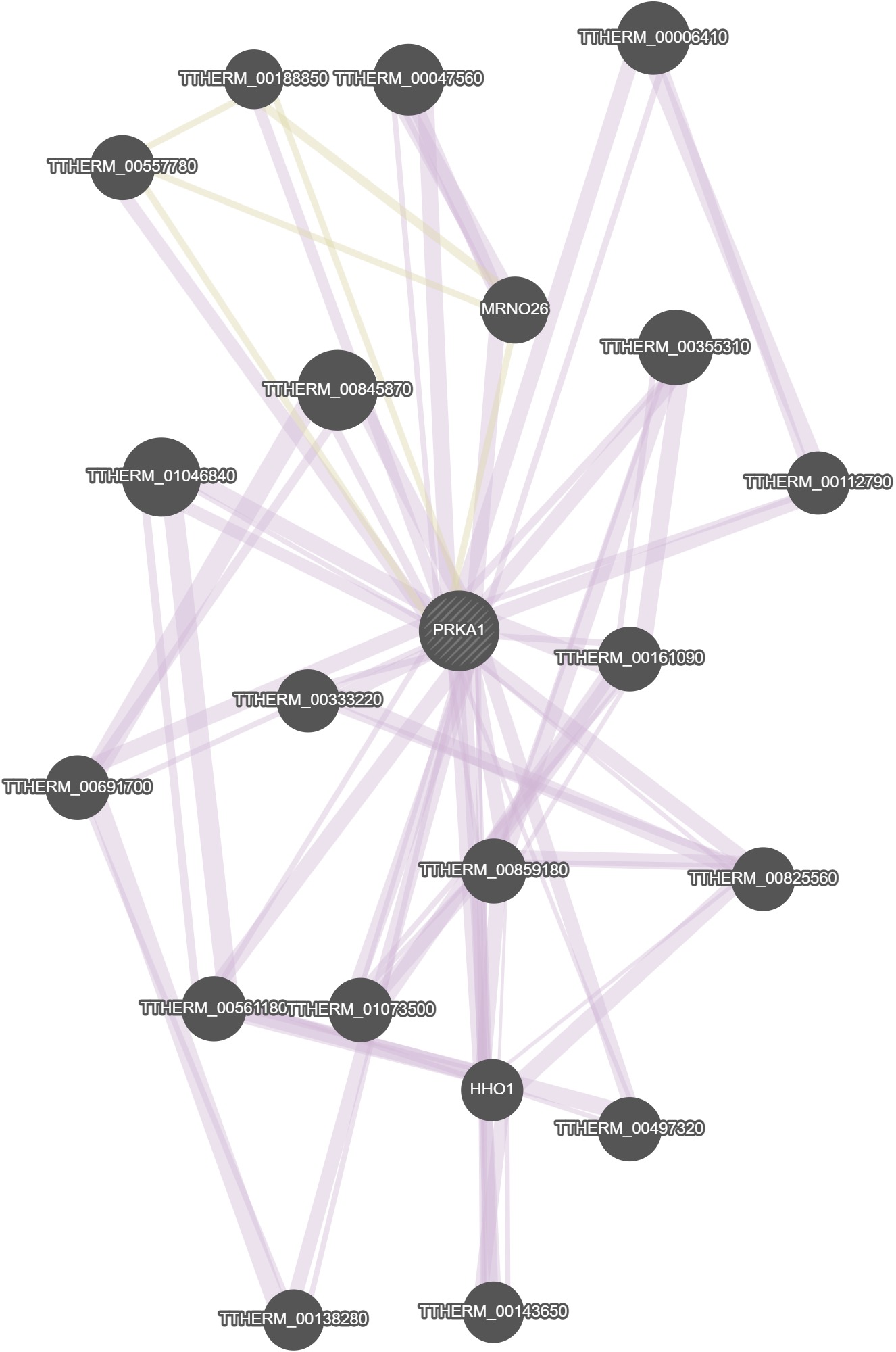
No results were calculated for this gene in GeneMania.
 Tetrahymena Stock Center
Tetrahymena Stock Center
No Data fetched for Tetrahymena Stock Center
 Homologs
Homologs
No Data fetched for Homologs
 General Information
General Information
No Data fetched for General Information
 Associated Literature
Associated Literature
- Ref:16933976: Eisen JA, Coyne RS, Wu M, Wu D, Thiagarajan M, Wortman JR, Badger JH, Ren Q, Amedeo P, Jones KM, Tallon LJ, Delcher AL, Salzberg SL, Silva JC, Haas BJ, Majoros WH, Farzad M, Carlton JM, Smith RK, Garg J, Pearlman RE, Karrer KM, Sun L, Manning G, Elde NC, Turkewitz AP, Asai DJ, Wilkes DE, Wang Y, Cai H, Collins K, Stewart BA, Lee SR, Wilamowska K, Weinberg Z, Ruzzo WL, Wloga D, Gaertig J, Frankel J, Tsao CC, Gorovsky MA, Keeling PJ, Waller RF, Patron NJ, Cherry JM, Stover NA,
 Sequences
Sequences
>TTHERM_00823550(coding)
ATGACAAGTACAAATAGCGGAAACACATTAAAAATTGAGCATTATCTTGTAGGAAAAACA
TTAGGGTGCGGAGCTTCTGGAAAAGTTAAGCTTGCCAAGCATGATATAACTGGAAAAGAA
GTTGCAATAAAAATTATAAATAAGAAAAAAATGACTGTTAAAAAAATGAGCAACAAGATT
AAAAGAGAAATTAGGCTTTTAAGATTCTTCAACCATTAAAACATTATTCGTTTATATGAA
GTTCTTGATACTAATACTGATATTTTTGTGGTTACTGAGTATATTTCAGGGGGAGATTTA
TATGATGTTATTGCTTCAAAGGGTAAGCTTCCTGAATAAGAAGCAAAAAGATATTTTAAG
TAAATAGTAGCAGGAGTTGATTACTGCCATAGAAACTTAGTCGCCCATAGAGATCTGAAA
CTTGAAAATATATTAATAGATGATAATAATAACATTAAAATTGCAGATTTTGGTTTGAGT
AATATTATGAATGATGGTAAATATTTGTCTACATCTTGTGGTTCTCCTAATTACGCTGCC
CCAGAAGTTATATCTGGAAAACTATATTGTGGAACAGAAGTTGATACTTGGAGTTGTGGT
GTTATATTATTCGCCTTGCTAGGAGGCTATCTTCCTTTTGATGAAGAAGTTATTCCAGCT
TTGTATAAGAAAATCAAGGAGGGAGATTTTCAGATTCCAGCATTTTTTTCACCTGAAGCT
CATGATATAATTAAAAGAATGCTCCGTCCTAACCCTATAGAGAGAATTAAATTTCATGAA
TTGAGACTTCATCCTTGGTTGAGAGAAAATGTTCCCTTTTATGTAGAAATATTCAATTAG
AATACAAGAATGGAGAGTAAAAAATTAAATGAAGACATTTTGAGAAAATTAGCTTAAATG
AAAAATGTTAACTTCCATAATTTGAACGAATAAACTATTAGAGATGCCGTTATTAAGAGA
GACGATTATAGTTTTGTTATTGCTTACGATTTGCTCCTTGATGACTATAAAAAGAACAAC
TATTATAATAAAGAAAACAATCAAAACACTTTCATATTCAGACCTGTTTAAGAAGACTGC
ACAGTCGAGACCAATAATGCTTATATTGATCAAATCTTTAATCAAGGAAAACTTGAAGAA
GTAACTGAGGAAGAAGAAGAAATACTTGGCCAGTGGACTTATGGAAAGATATATAGCAAT
AGTGCTAGGACAATTATGAAAGCAATATATGAGGTTGCAAGCTAGTTAGATATTGTTATT
ACAATTAAAAATAGCGAATATAGGATCAAATGCCATAGAAAAAACCCTAAAAAGATCTAA
AGTGCTTACAACAACCATCTTGAAACTATAAAAAGTGAAAGTAGCTCAAATTTATCAACA
AATTACACAAATAATGGTAATTTAAACAACAGCCACCACCATCACCACTCATACAATAAT
CATTACCATCATGTTAATCCATAGAATTAAAATTTTACTGGCTTCTAGAGTGGATCAAAT
GTTTCTATGACAAACAACTTTTAGCACACATAAACTAATTCCTATTTAATGCCTCCCTAA
GGTAATTAATTTAATAATTAAATGAGTAATGTAAGCAATGACTTTGGATAAAATATGGAA
TCTCAATCTTAATCTTATGCCATAGGTAGCAATTTCAATAATATGAATTATAGTCAATCG
GGATATTAATTTGATTCTGGAATGAATCTAAATAATGGTAACACTTAAAACCCAAATAAC
AGTTAAAATAATGCTCTTGACACTAATAACATACCTAAATTCTCCAAATAGGATCTTGTT
TTTACCATATAAATATACTAAATGCCAAGCACTTCAATTCAAAGCGATTAGCACATAGTA
GATGTCATTTTAAATAAGGGTCATCCTATCATGTTCTTAGAGTTTTTTAACTAATTTCTT
ACTCTTGTAAACAAAAAACTTAATCCAATTCACTGA
>TTHERM_00823550(gene)
AAACAAATTAAAAGTAAATATTAATTAATACATTAAATAAAAGCAAGTTATTAAAATGAC
AAGTACAAATAGCGGAAACACATTAAAAATTGAGCATTATCTTGTAGGAAAAACATTAGG
GTGCGGAGCTTCTGGAAAAGTTAAGCTTGCCAAGCATGATATAACTGGAAAAGAAGTTGC
AATAAAAATTATAAATAAGAAAAAAATGACTGTTAAAAAAATGAGCAACAAGATTAAAAG
AGAAATTAGGCTTTTAAGATTCTTCAACCATTAAAACATTATTCGTTTATATGAAGTTCT
TGATACTAATACTGATATTTTTGTGGTTACTGAGTATATTTCAGGGGGAGATTTATATGA
TGTTATTGCTTCAAAGGGTAAGCTTCCTGAATAAGAAGCAAAAAGATATTTTAAGTAAAT
AGTAGCAGGAGTTGATTACTGCCATAGAAACTTAGTCGCCCATAGAGATCTGAAACTTGA
AAATATATTAATAGATGATAATAATAACATTAAAATTGCAGATTTTGGTTTGAGTAATAT
TATGAATGATGGTAAATATTTGTCTACATCTTGTGGTTCTCCTAATTACGCTGCCCCAGA
AGTTATATCTGGAAAACTATATTGTGGAACAGAAGTTGATACTTGGAGTTGTGGTGTTAT
ATTATTCGCCTTGCTAGGAGGCTATCTTCCTTTTGATGAAGAAGTTATTCCAGCTTTGTA
TAAGAAAATCAAGGAGGGAGATTTTCAGATTCCAGCATTTTTTTCACCTGAAGCTCATGA
TATAATTAAAAGAATGCTCCGTCCTAACCCTATAGAGAGAATTAAATTTCATGAATTGAG
ACTTCATCCTTGGTTGAGAGAAAATGTTCCCTTTTATGTAGAAATATTCAATTAGAATAC
AAGAATGGAGAGTAAAAAATTAAATGAAGACATTTTGAGAAAATTAGCTTAAATGAAAAA
TGTTAACTTCCATAATTTGAACGAATAAACTATTAGAGATGCCGTTATTAAGAGAGACGA
TTATAGTTTTGTTATTGCTTACGATTTGCTCCTTGATGACTATAAAAAGAACAACTATTA
TAATAAAGAAAACAATCAAAACACTTTCATATTCAGACCTGTTTAAGAAGACTGCACAGT
CGAGACCAATAATGCTTATATTGATCAAATCTTTAATCAAGGAAAACTTGAAGAAGTAAC
TGAGGAAGAAGAAGAAATACTTGGCCAGTGGACTTATGGAAAGATATATAGCAATAGTGC
TAGGACAATTATGAAAGCAATATATGAGGTTGCAAGCTAGTTAGATATTGTTATTACAAT
TAAAAATAGCGAATATAGGATCAAATGCCATAGAAAAAACCCTAAAAAGATCTAAAGTGC
TTACAACAACCATCTTGAAACTATAAAAAGTGAAAGTAGCTCAAATTTATCAACAAATTA
CACAAATAATGGTAATTTAAACAACAGCCACCACCATCACCACTCATACAATAATCATTA
CCATCATGTTAATCCATAGAATTAAAATTTTACTGGCTTCTAGAGTGGATCAAATGTTTC
TATGACAAACAACTTTTAGCACACATAAACTAATTCCTATTTAATGCCTCCCTAAGGTAA
TTAATTTAATAATTAAATGAGTAATGTAAGCAATGACTTTGGATAAAATATGGAATCTCA
ATCTTAATCTTATGCCATAGGTAGCAATTTCAATAATATGAATTATAGTCAATCGGGATA
TTAATTTGATTCTGGAATGAATCTAAATAATGGTAACACTTAAAACCCAAATAACAGTTA
AAATAATGCTCTTGACACTAATAACATACCTAAATTCTCCAAATAGGATCTTGTTTTTAC
CATATAAATATACTAAATGCCAAGCACTTCAATTCAAAGCGATTAGCACATAGTAGATGT
CATTTTAAATAAGGGTCATCCTATCATGTTCTTAGAGTTTTTTAACTAATTTCTTACTCT
TGTAAACAAAAAACTTAATCCAATTCACTGAGTTTCAGTTTGATGCTCAAGCTAATTTCT
TTTAATTAAAACTTTAAACATTTAAAATTGTAAATTCATATTTTTGTATTCTTTGGTAAA
GCAAAAATAAATTAAATAAATTCAT
>TTHERM_00823550(protein)
MTSTNSGNTLKIEHYLVGKTLGCGASGKVKLAKHDITGKEVAIKIINKKKMTVKKMSNKI
KREIRLLRFFNHQNIIRLYEVLDTNTDIFVVTEYISGGDLYDVIASKGKLPEQEAKRYFK
QIVAGVDYCHRNLVAHRDLKLENILIDDNNNIKIADFGLSNIMNDGKYLSTSCGSPNYAA
PEVISGKLYCGTEVDTWSCGVILFALLGGYLPFDEEVIPALYKKIKEGDFQIPAFFSPEA
HDIIKRMLRPNPIERIKFHELRLHPWLRENVPFYVEIFNQNTRMESKKLNEDILRKLAQM
KNVNFHNLNEQTIRDAVIKRDDYSFVIAYDLLLDDYKKNNYYNKENNQNTFIFRPVQEDC
TVETNNAYIDQIFNQGKLEEVTEEEEEILGQWTYGKIYSNSARTIMKAIYEVASQLDIVI
TIKNSEYRIKCHRKNPKKIQSAYNNHLETIKSESSSNLSTNYTNNGNLNNSHHHHHSYNN
HYHHVNPQNQNFTGFQSGSNVSMTNNFQHTQTNSYLMPPQGNQFNNQMSNVSNDFGQNME
SQSQSYAIGSNFNNMNYSQSGYQFDSGMNLNNGNTQNPNNSQNNALDTNNIPKFSKQDLV
FTIQIYQMPSTSIQSDQHIVDVILNKGHPIMFLEFFNQFLTLVNKKLNPIH
 Identifiers and Description
Identifiers and Description
 External Links
External Links
 Gene Ontology Annotations
Gene Ontology Annotations
 Domains
Domains
 Gene Expression Profile
Gene Expression Profile
 Vegetative Cell Cycle (Zhang et al.,
2023)
Vegetative Cell Cycle (Zhang et al.,
2023)
 GeneMania
GeneMania
 Tetrahymena Stock Center
Tetrahymena Stock Center  Homologs
Homologs
 General Information
General Information
 Associated Literature
Associated Literature
 Sequences
Sequences



